LES VIRUS
Les virus sont universellement répandus dans le monde vivant mais sont incapables de se reproduire par eux-mêmes.
Pour assurer leur multiplication, ils sont totalement dépendants de cellules animales ou végétales, de champignons ou de bactéries, dont ils prennent le contrôle.
Ce contrôle est assuré par des instructions codées dans leur ADN ou leur ARN selon le type de virus. Tous les matériaux de base, la machinerie qui les assemble et l’énergie nécessaire à ces processus proviennent de l’organisme qu’ils infectent.
Les virus se présentent sous deux formes, nue ou enveloppée.
La forme nue est formée d’ARN ou d’ADN associé à des protéines pour former une capside. Chez les virus enveloppés, ces capsides sont entourées par une double couche de lipides. Le SARS-CoV2 est un virus enveloppé à ARN.
Virus nu Virus enveloppé
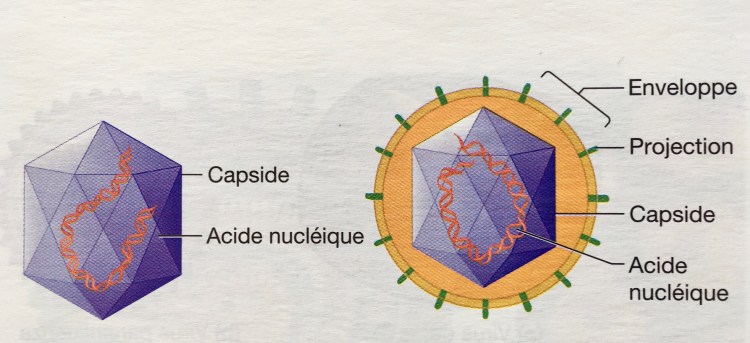
Prescott et al., Microbiologie, 3ème édition, 2010, Editions De Boek
Chez le SARS-CoV-2, la couche de lipides est constituée par un fragment de membrane « emprunté » à la cellule infectée lors de l’assemblage final du virus. De cette enveloppe émerge une couronne de pics appelés spicules (S).
Structure d’un coronavirus
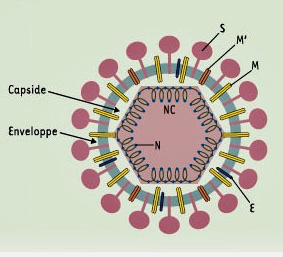
Le « ressort » présent à l’intérieur de la capside correspond à l’ARN.
En microscopie électronique, le SARS-CoV-2 apparaît sous forme de particules sphériques appelées virions, d’un diamètre de 60 à 140 nanomètres (1 nm = 1 milliardième de m). Les spicules sont répartis en couronne à la surface du virion et mesurent entre 9 et 12 nm.
Le virion n’est pas visible à l’intérieur de la cellule hôte, car il s’y fixe extérieurement par l’intermédiaire des spicules, mais n’y pénètre pas. Il perd son enveloppe lors de la fusion avec les membranes cellulaires. Seule pénétrera la capside contenant l’ARN.
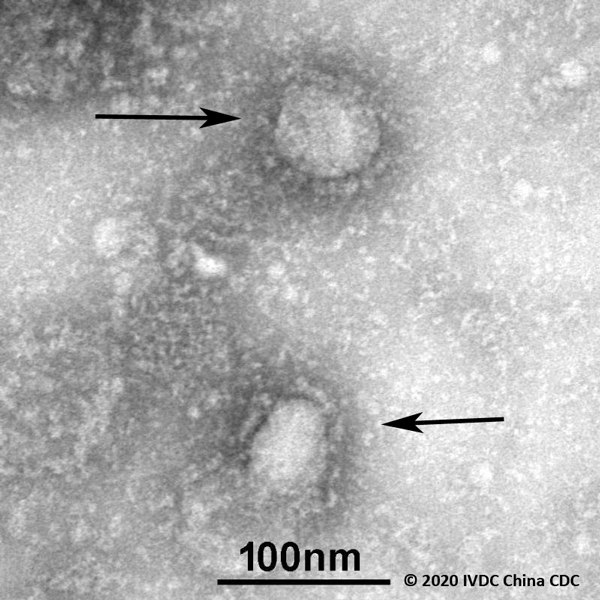
Visualisation de virions du SARS-CoV-2 en microscopie électronique
https://www.nejm.org/doi/pdf/10.1056/NEJMoa2001017?articleTools=true
ARN et ADN : les donneurs d’ordres du vivant
Tous les êtres vivants, sans exception, se développent à partir d’un programme spécifique, présent dans leur ARN ou leur ADN.
Dans le cas des coronavirus, ce programme est stocké sous forme d’ARN.
L’ARN (Acide Ribo Nucléique) : Que recouvre cette appellation ?
Acide : l’ARN est dit acide car il libère des ions hydrogène (H+).
Ribo : abréviation de Ribose. En biochimie, le suffixe « ose » est très souvent employé pour nommer un sucre. La première partie du mot peut désigner la provenance. Il en est ainsi dans « fructose », un sucre provenant des fruits ou dans « lactose », un sucre provenant du lait.
Dans le cas du ribose, l’origine du mot est tout autre. Ce sucre a été découvert en 1908 par un biochimiste et un chimiste qui travaillaient à New York, au Rockefeller institute of biochemistry, d’où le préfixe Rib.
Le ribose présent dans l’ARN contient cinq atomes de carbone. Il peut être schématisé de la façon suivante :
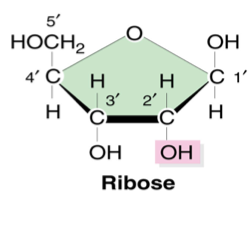
Nucléique : L’ARN a une structure très proche de celle de l’ADN. Ce dernier ayant été appelé acide nucléique, car mis originellement en évidence dans les noyaux cellulaires, la dénomination « nucléique » a été étendue à l’ARN.
Structure de l’ARN
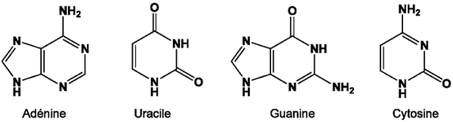
L’appellation ARN, que nous venons de détailler, ne permet pas à elle seule de comprendre la structure de l’ARN. Outre l’acide (sous forme de groupements phosphates) et le sucre (ribose), on trouve également quatre bases que l’on abrège par les lettres A, U, G, C :
L’association du ribose, de groupes phosphates et d’une base constitue un nucléotide :
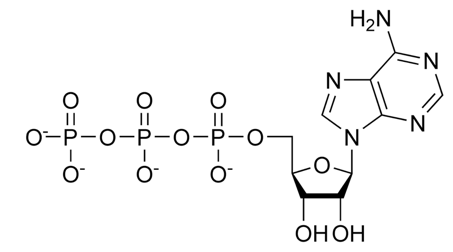
Exemple de nucléotide de l’ARN avec l’adénine comme base
L’ARN est constitué par un enchaînement de ces nucléotides.
Le sucre (ribose) et le phosphate forment un « squelette » auquel sont accrochées les bases.
Exemple d’enchaînement de quatre nucléotides :
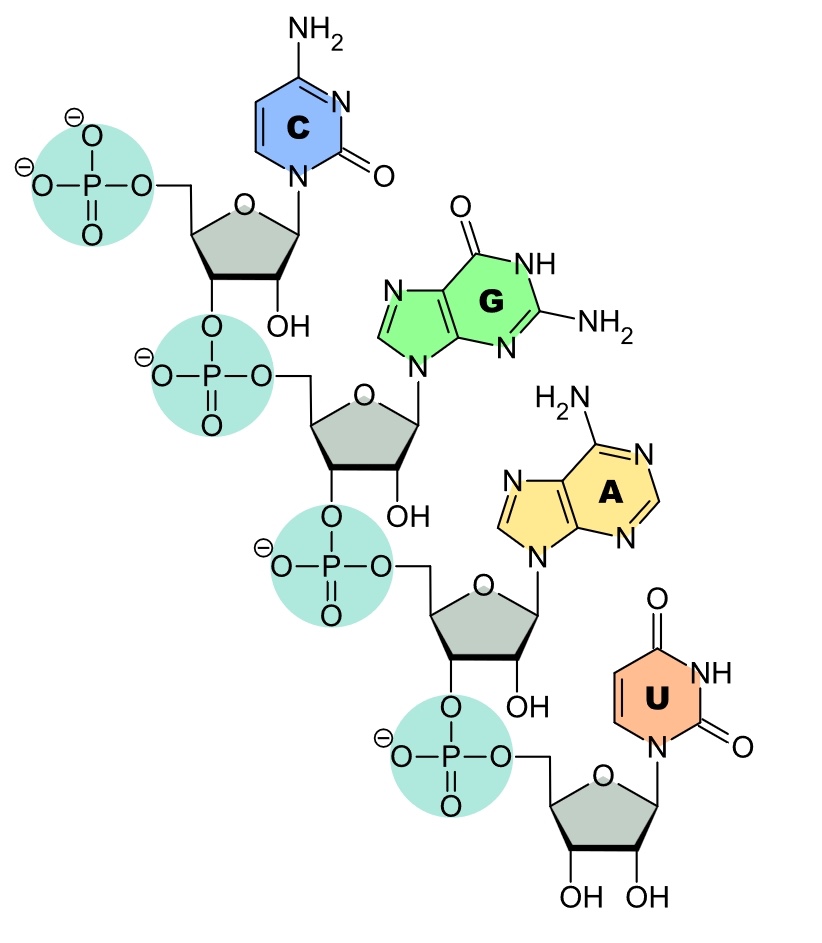
L’ordre d’enchaînement des bases le long de ce squelette définit la séquence de l’ARN. Pour représenter commodément ces séquences, on décrit la succession des bases sous leur forme abrégée, par exemple : CCCACAGCCGCCAGUUCCGCUGGCGGCAUUUU
L’un des premiers SARS-CoV-2 séquencé provenait d’un patient qui travaillait sur le marché de Wuhan. Cet ARN était constitué de 29 903 nucléotides répartis comme indiqué dans le schéma ci-dessous.
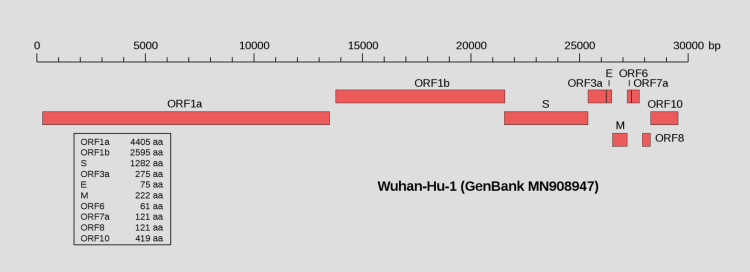
Disposition des gènes le long de l’ARN du SARS-CoV-2
https://www.nature.com/articles/s41586-020-2008-3
Sur ce schéma, on remarque la présence de plusieurs rectangles rouges. Chacun de ces rectangles correspond à un gène du SARS-CoV-2. Un gène est constitué par une séquence de plusieurs nucléotides.
A cette séquence de nucléotides correspond une séquence de plusieurs acides aminés, c’est à dire une protéine.
Prenons pour exemple le gène S, ainsi appelé car il porte l’information pour élaborer la protéine du spicule.
Ce gène s’étend du nucléotide 21563 au nucléotide 25384. Comme indiqué dans le tableau situé à gauche du schéma, il correspond à une protéine de 1282 acides aminés (aa).
L’évolution de ce gène est surveillée de très près, car le virion se fixe aux cellules par le spicule. Si l’extrémité du spicule est modifiée, il en sera de même pour l’ancrage du virus.
Suivant les modifications de l’extrémité du spicule, cet ancrage pourra être plus important ou plus faible, voire pratiquement inexistant. Dans ce dernier cas, le virus ne sera plus infectieux.
Notion de mutation
Lors de la multiplication des virus à ARN, des changements fréquents interviennent dans la séquence des nucléotides par rapport à l’ARN originel. Ces modifications sont appelées mutations.
Cette propension à muter est due en partie à l’absence de mécanismes permettant de localiser et de réparer les séquences erronées qui apparaissent fréquemment lors de la copie de l’ARN.
Les virus à ADN sont beaucoup plus stables car ils bénéficient des systèmes de réparation très sophistiqués dont disposent les cellules hôtes pour la reproduction de leur propre ADN.
Afin de détecter les mutations du SARS-CoV2, plusieurs centaines d’analyses de séquences sont réalisées chaque jour, dans différents pays, à partir de prélèvements effectués sur des patients. Les résultats sont en accès direct sur https://nextstrain.org/ncov/global et permettent à chacun de suivre l’évolution du virus.
On obtient ainsi un arbre montrant les relations de parenté entre les différents SARS-CoV-2 analysés. Il s’agit d’un « arbre généalogique », construit à partir de séquences génétiques :

Un point coloré correspond au séquençage d’un virus complet. En allant sur le site et en cliquant sur l’un d’eux, on peut observer la présence éventuelle de mutations et obtenir le détail de celles-ci. Les couleurs diffèrent selon le pays d’origine.
Dans la carte « transmissions » située sur le même site, les données génétiques sont reliées aux données géographiques et permettent de visualiser la dynamique de la pandémie.
Notion de recombinaison
Un autre processus permet de modifier de façon importante les séquences de nucléotides chez les virus à ARN, il s’agit de la recombinaison de segments de gènes.
Ce processus est notamment illustré par l’évolution, durant près de 90 ans, du virus H1N1 de la grippe dite « espagnole » de 1918, qui est en fait partie des USA et qui a abouti à la pandémie du H1N1 de 2009. https://academic.oup.com/emph/article/2019/1/18/5298310
Relations génétiques entre les virus des grippes humaines et porcines, 1918-2009
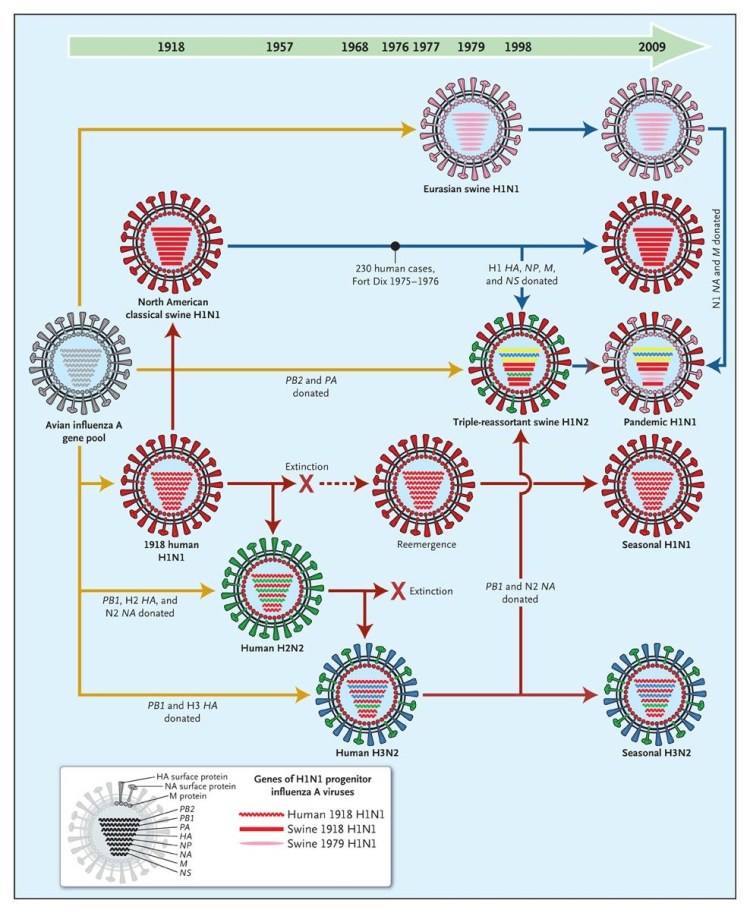
https://www.nejm.org/doi/pdf/10.1056/NEJMp0904819?articleTools=true
https://www.nejm.org/doi/pdf/10.1056/NEJMra0904322?articleTools=true
Le virus H1N1 présent chez les humains décédés de cette épidémie en 1918 a été récupéré à partir de deux sources :
- Des tissus pulmonaires de malades conservés dans le formol après autopsie
- Des poumons prélevés chez une femme originaire d’Alaska et inhumée dans le permafrost, donc congelée.
Il en ressort que depuis 1918, ce virus initialement présent chez les oiseaux a circulé sans discontinuer entre les espèces porcines et humaines. Qui plus est, en 1918, il a été transmis par l’homme à l’espèce porcine.
Cet exemple illustre les sauts d’espèces qui ont lieu en permanence et qui, d’ailleurs, ne sont pas propres au seul virus de la grippe.
SARS-CoV2 et saut d’espèce
Comme précédemment, il est possible de comparer les séquences du SARS-CoV-2 prélevé chez l’homme avec celles d’autres coronavirus prélevés chez les animaux.

R. Lu et Coll., Lancet 2020 ;395 : 565-74
Cet arbre phylogénétique montre que les virus SARS-CoV-2 (notés 2019-nCoV) trouvés chez l’homme sont très proches de ceux trouvés chez certaines chauve-souris (Bat-SL-CoV C45 et C21). L’analogie sur l’ensemble de leurs gènes est de l’ordre de 88%.
https://www.thelancet.com/journals/lancet/article/PIIS0140-6736(20)30251-8/fulltext
https://royalsocietypublishing.org/doi/pdf/10.1098/rstb.2004.1478
Virus à ARN : Un brassage génétique permanent
Ces brassages génétiques par mutations et recombinaisons sont une des clefs du « succès » des virus à ARN et expliquent, en partie, leur propension à coloniser l’ensemble du monde vivant.
De fait, on constate qu’un grand nombre de maladies virales auxquelles l’homme doit faire face, et qui peuvent aboutir à des pandémies, sont dues à des virus à ARN : HIV, Ebola, Chikungunya, Zika, SARS-CoV, dengue, poliomyélite, rage, grippes, rhumes, rougeole, rubéole, oreillons, fièvre jaune, hépatites A et C.
De même, la majorité des virus pathogènes pour les plantes sont des virus à ARN.
https://fr.wikipedia.org/wiki/Liste_de_phytovirus
https://apsjournals.apsnet.org/doi/pdfplus/10.1094/PHYTO-07-18-0257-RVW
Cependant, tous les virus à ARN ne sont pas pathogènes, et une grande majorité d’entre eux, ainsi que certains virus à ADN, jouent un rôle fondamental dans l’équilibre des organismes vivants.
Il faut souligner que ces approches moléculaires de l’évolution des êtres vivants ont été rendues possibles par la révolution technologique en matière de séquençage à haut débit et en bioinformatique.
Fixation du SARS-CoV-2 sur les cellules
Comme précédemment indiqué, le gène S possède l’information nécessaire pour piloter la synthèse de la protéine des spicules.
Cette protéine comprend différents domaines et notamment un domaine de liaison, coloré en vert dans le schéma ci-dessous :
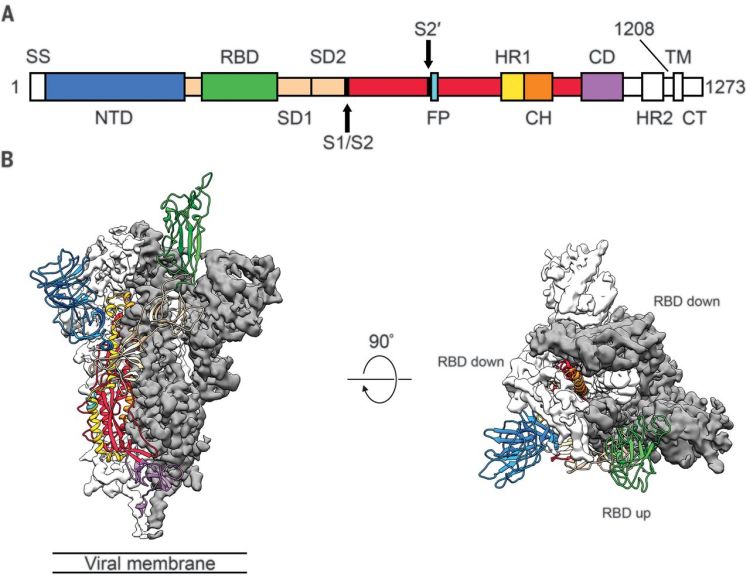
Ce domaine RBD (Receptor Binding Domain = domaine de liaison au récepteur cellulaire), situé à l’extrémité du spicule, permet l’ancrage des virus sur les récepteurs présents à la surface de la cellule hôte. Lorsque le spicule est représenté en 3D (Partie B du schéma), le domaine coloré en vert, en forme de « spaghetti », correspond à cette zone d’ancrage.
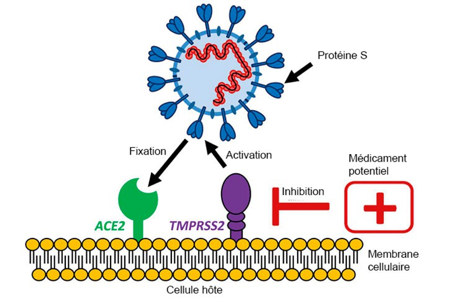
Les extrémités des spicules (domaine RBD) se fixent sur un récepteur appelé ACE2 (Angiotensin Converting Enzyme 2) ou enzyme de conversion de l’angiotensine, situé à la surface des cellules. Par rapport au SARS-CoV1 de 2003, l’affinité du SARS-CoV2 pour ce récepteur est de 10 à 20 fois plus importante.
https://science.sciencemag.org/content/367/6483/1260/tab-pdf
Un grand nombre de récepteurs ACE2 sont situés au niveau des poumons, à la surface des cellules alvéolaires de type II qui sécrètent le surfactant pulmonaire.
Outre ce foyer pulmonaire, nombre de récepteurs ont été mis en évidence au niveau de l’épithélium de l’intestin grêle.
Enfin, ces récepteurs sont également présents à la surface des cellules endothéliales et des muscles lisses des artères et des veines de la plupart des organes (foie, reins, cerveau, rate, etc.).
https://onlinelibrary.wiley.com/doi/full/10.1002/path.1570
Le récepteur ACE2 fait partie intégrante des cellules hôtes et agit comme un « ciseau moléculaire » :
Il coupe très faiblement l’angiotensine I biologiquement inactive et la transforme en angiotensine II active.
Il coupe principalement l’angiotensine II active et la transforme en angiotensine inactive.
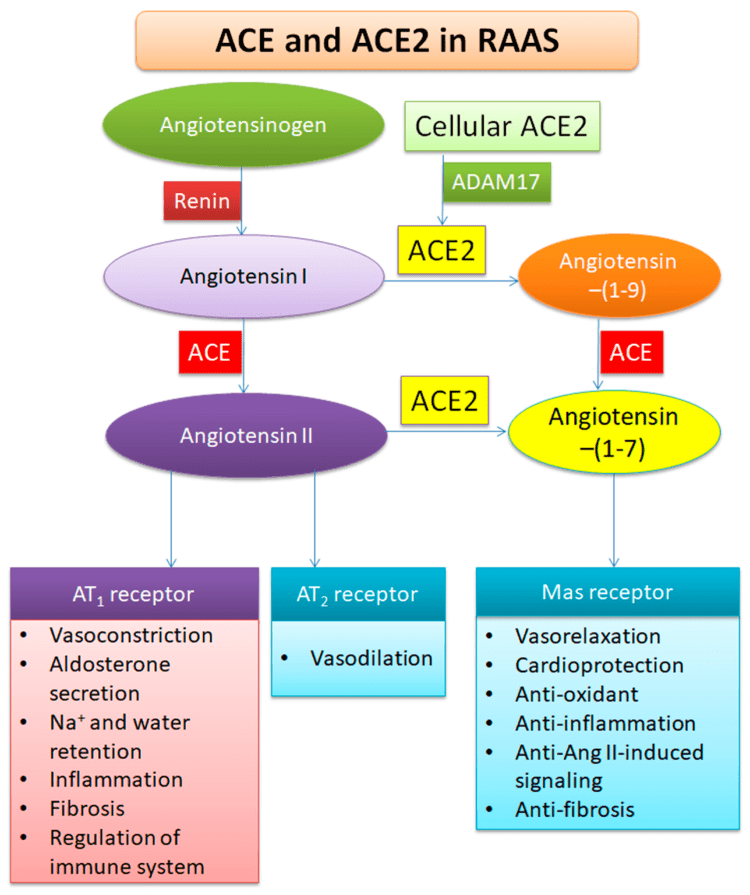
Ainsi, le virion du SARS-CoV-2 se fixe sur les cellules de notre organisme via un important élément régulateur de la pression artérielle.
Après fixation sur le récepteur ACE2, la fusion de l’enveloppe virale avec la membrane cellulaire nécessite une coupure de l’extrémité du spicule par d’autres « ciseaux moléculaires » (TMPRSS2) présents à la surface des cellules hôtes. Sans cette coupure, il ne peut y avoir de fusion.
c est très détaillé et très bien expliqué.
je vous félicite
super
J’aimeJ’aime