La PCR (Polymerase Chain Reaction ou réaction de polymérisation en chaîne) permet d’obtenir un très grand nombre de copies d’une séquence d’ADN préalablement sélectionnée
PCR : Une amplification sélective de l’ADN
Des sources chaudes à la PCR
Dans les années 1960 une majorité de scientifiques était convaincue que les organismes vivants ne pouvaient supporter des températures supérieures à 70°C.
En 1969, le microbiologiste américain Thomas Brock et son jeune assistant Hudson Freeze révèlent la présence, dans les sources chaudes du Parc de Yellowstone aux États-Unis, d’une bactérie supportant ces températures. Ils la nomment Thermus aquaticus.

En 1976, une équipe de biochimistes et de biologistes moléculaires de l’Université de Cincinnati purifie une ADN polymérase à partir de cette bactérie. Une ADN polymérase est une enzyme qui enchaîne les nucléotides (Bases A,T,G ou C + sucre + triphosphates) pour former un nouveau brin d’ADN en copiant un brin d’ADN déjà existant. Cette enzyme, qui sera ultérieurement appelée Taq polymérase (le terme Taq correspondant au diminutif de Thermus aquaticus), fonctionne de façon optimale à 80°C et résiste à des températures autour de 95°C.
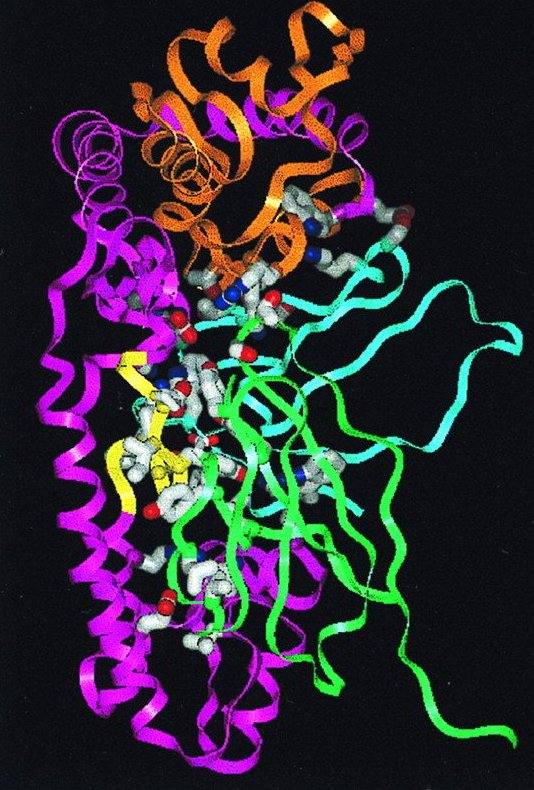
Structure 3D de la Taq polymérase, PNAS 1998
De la Taq polymérase à la PCR …
Jusqu’au milieu des années 1980, la multiplication (aussi appelée amplification ou clonage) de séquences spécifiques d’ADN nécessitait l’utilisation de méthodes longues et laborieuses. Il fallait tout d’abord isoler la région d’ADN à amplifier puis l’introduire dans une bactérie. Après multiplication des bactéries, il fallait extraire et purifier les séquences d’ADN amplifiées.
En 1985, le biochimiste américain Kary Mullis publie dans Science une méthode d’amplification de l’ADN beaucoup plus simple. Elle est basée sur l’utilisation dans un tube à essai d’une ADN polymérase extraite de la bactérie Escherichia coli et de fragments d’ADN de synthèse, les amorces, encadrant la zone à amplifier.
Un problème subsistait néanmoins avec cette méthode, à savoir le manque de résistance de l’ADN polymérase d’Escherichia coli à la chaleur. Ce problème fût surmonté en 1988 en utilisant la Taq polymérase.
Kary Mullis reçut le prix Nobel de chimie en 1993 « Pour son invention de la méthode de réaction en chaîne de la polymérase » connue sous l’abréviation PCR (Polymerase Chain Reaction).

Kary Mullis (1944-2019), Prix Nobel de chimie 1993
Cette méthode, qui permet d’obtenir de multiples copies d’une séquence choisie d’ADN, fut universellement adoptée par les biologistes moléculaires et fait actuellement partie intégrante de leur boîte à outils.
En biologie médicale, les applications de cette technique sont multiples et concernent les modifications génétiques liées aux cancers, le diagnostic prénatal non invasif, les maladies génétiques ainsi que la détection des infections bactériennes ou virales.
En police scientifique, la PCR est utilisée au quotidien pour l’identification des personnes. Dans ce cas, elle est communément appelée « analyse ADN » ou analyse « d’empreintes génétiques ». Elle est également employée pour les analyses de paternité.
Principe de base de la PCR
Préalablement à la mise au point d’une analyse par PCR, il faut avoir séquencé la zone de l’ADN à amplifier, c’est à dire avoir déterminé l’ordre de succession des bases (A,T,G,C). Ce séquençage permet de concevoir les amorces qui permettent l’amorçage de la réaction de polymérisation par la Taq polymérase. Séquençage et synthèse des amorces sont réalisés dans des laboratoires spécialisés.
Les laboratoires d’analyse peuvent ainsi disposer d’amorces spécifiquement conçues pour leurs différentes problématiques, qu’il s’agisse de biologie médicale ou de police scientifique.
Une analyse par PCR comporte plusieurs cycles successifs, constitués chacun de trois étapes identiques de changement de température.
La première étape consiste à ouvrir la double hélice d’ADN en portant la température du milieu à 95°C.
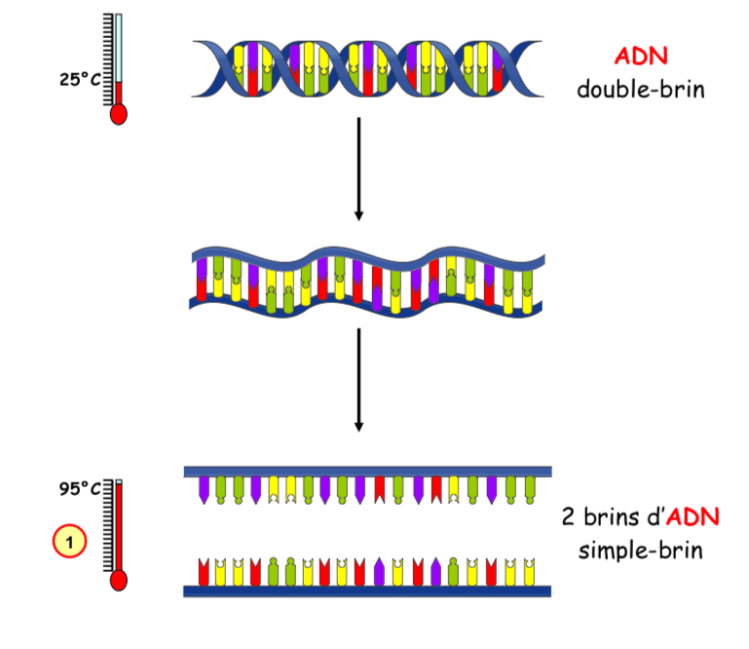
Phase 1 : Ouverture de la double hélice d’ADN
Une fois la double hélice ouverte, la deuxième étape consiste à abaisser la température du milieu pour permettre la fixation d’une paire d’amorces de synthèse sur les brins d’ADN. Les amorces sont constituées d’une séquence d’une vingtaine de bases (A,T,G,C) qui vont encadrer la zone d’ADN à amplifier. Cette étape de fixation des amorces est appelée hybridation.
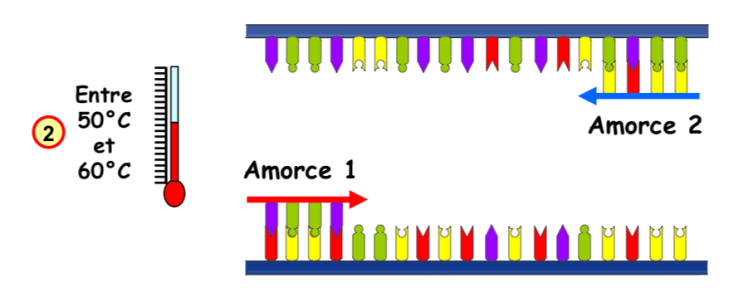
Phase 2 : Fixation des amorces (Hybridation)
Au cours de la troisième étape la température du milieu réactionnel est portée à 72°C pour permettre l’action de la Taq polymérase. Durant cette étape les bases (A,T,G,C,), ajoutées dans le milieu sous forme de nucléotides, se positionnent devant les bases correspondantes des zones à amplifier. Les nucléotides sont ensuite reliés entre eux pour former un nouveau brin d’ADN.
Dans le schéma ci-dessous les 4 types de nucléotides sont désignés par l’abréviation dNTPs(désoxyribo Nucléosides Tri Phosphates).
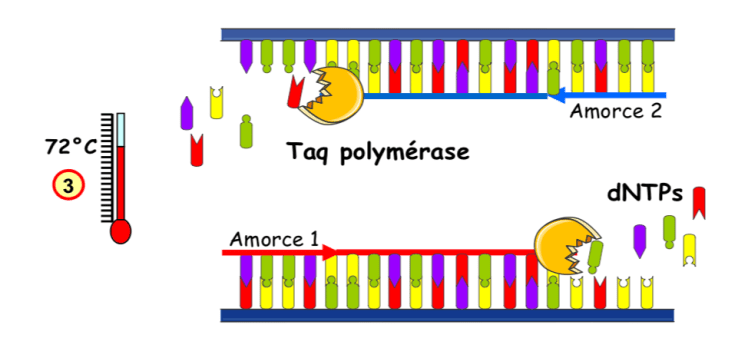
Phase 3 : Synthèse d’ADN par la Taq polymérase (Etape dite d’élongation)
Les changements de température correspondant à ces trois étapes sont réalisés avec un thermocycleur. Ces appareils contiennent des porte-échantillons métalliques couplés à un logiciel permettant de piloter les températures, les temps de chauffe et le nombre de cycles à effectuer.

Thermocycleur : Permet la programmation des étapes de température et du nombre de cycles
Les échantillons à analyser sont placés dans des plaques de 96 ou de 384 puits qui font office de « tubes à essai » et qui reposent sur le porte échantillons du thermocycleur.

Plaques pour 96 ou 384 échantillons
Les trois étapes précédemment décrites vont être reproduites à l’identique au cours des cycles successifs de la PCR.
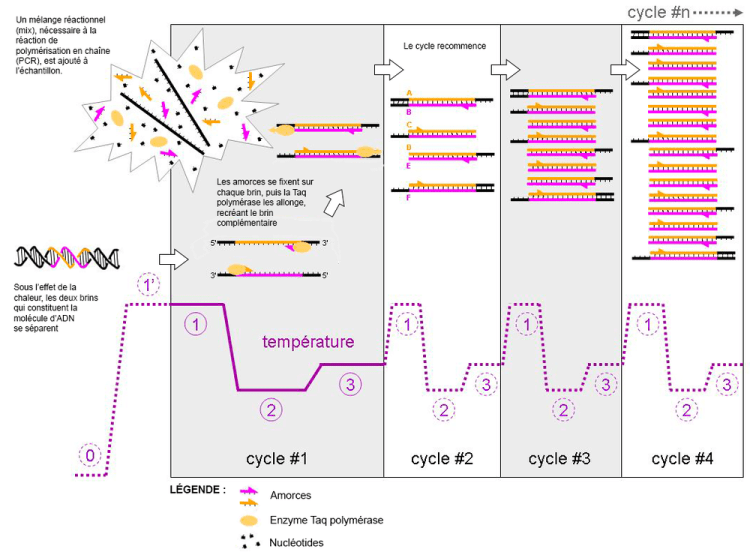
A partir du troisième cycle, deux copies de la zone à amplifier sont obtenues. Le nombre de copies à amplifier va ensuite doubler à chaque cycle supplémentaire.
Afin de visualiser l’amplification de l’ADN les amorces sont couplées à des sondes munies de marqueurs fluorescents. Chaque fois que de nouvelles copies sont créées, les sondes émettent un signal fluorescent. Plus le nombre de copies augmente, plus l’intensité de fluorescence augmente.
Comme présenté dans le graphe ci-dessous, pour 20 cycles, l’amplification sera de un million et pour 30 cycles elle avoisinera un milliard de copies.
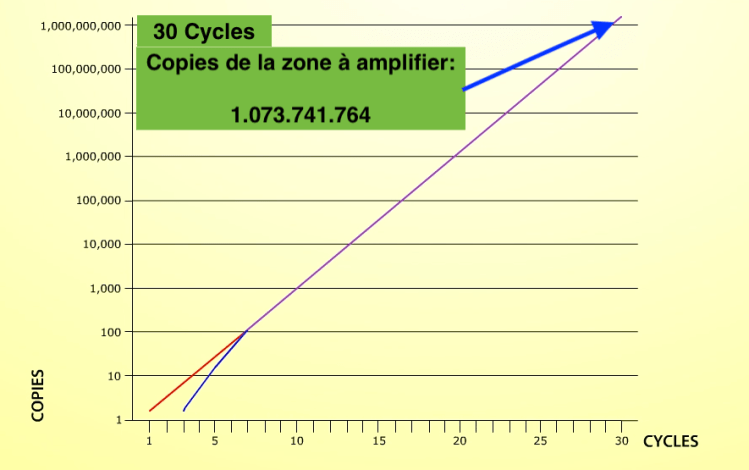
Nombre de copies en fonction du nombre de cycles
RT-PCR (Reverse Transcriptase PCR)
Le coronavirus Sars-Cov2 est un virus à ARN. Son identification par PCR nécessite donc une transformation préalable de séquences cibles de cet ARN en ADN. Cette transformation, dénommée transcription inverse, est réalisée avec une enzyme particulière, la transcriptase inverse.
Deux chercheurs américains, Howard Temin et David Baltimore, ont découvert cette enzyme, l’un à partir du virus du sarcome de Rous et l’autre à partir d’un virus de la souris. Le virus du sarcome de Rous induit chez le poulet des tumeurs qui présentent un caractère transmissible. Son découvreur, l’américain Francis Peyton Rous, put l’isoler et le cultiver dès 1911. Il reçut le prix Nobel en 1966 pour l’ensemble de ses travaux montrant le rôle des virus dans certains types de cancer.

Francis Peyton Rous (1879-1970)
En étudiant le virus du sarcome de Rous, Howard Temin s’aperçût qu’un composé inhibant la multiplication des virus à ADN empêchait sa croissance alors qu’il s’agit d’un virus à ARN. Il en déduisit que la multiplication de ce virus impliquait une phase pendant laquelle l’ARN viral était transformé en ADN afin de s’intégrer dans l’ADN de la cellule infectée. Il postula qu’il devait exister une enzyme capable de transformer l’ARN en ADN et découvrit la transcriptase inverse.
Un autre chercheur américain, David Baltimore, découvrit cette même enzyme chez un virus induisant des leucémies chez la souris. Leurs découvertes furent publiées le 27 juin 1970 dans le même numéro de Nature (1) (2). Ces virus à ARN qui synthétisent l’ADN via une transcriptase inverse sont appelés rétrovirus. Par ce biais, ils s’intègrent dans l’ADN des cellules infectées et peuvent être transmis à la descendance de ces dernières.
Depuis ces premières découvertes, de nombreux rétrovirus ont été mis en évidence chez les mammifères, dont le virus du SIDA (Syndrome d’Immuno Déficience Acquise) chez l’homme.

Utilisation de la transcriptase inverse en RT-PCR
Comme pour la PCR classique, la mise en route de la transcription inverse nécessite l’utilisation d’amorces d’ADN afin de délimiter la zone de l’ARN à amplifier. La fixation de ces amorces s’effectue autour de 55°C.
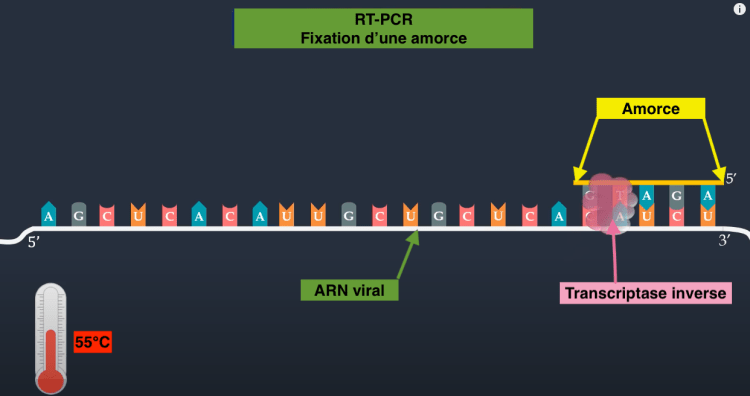
A partir de cette amorce d’ADN la transcriptase inverse va synthétiser un ADN complémentaire (ADNc) à partir de l’ARN viral. Les bases A de cet ADNc vont se positionner devant les bases U de l’ARN, et les G face au C.
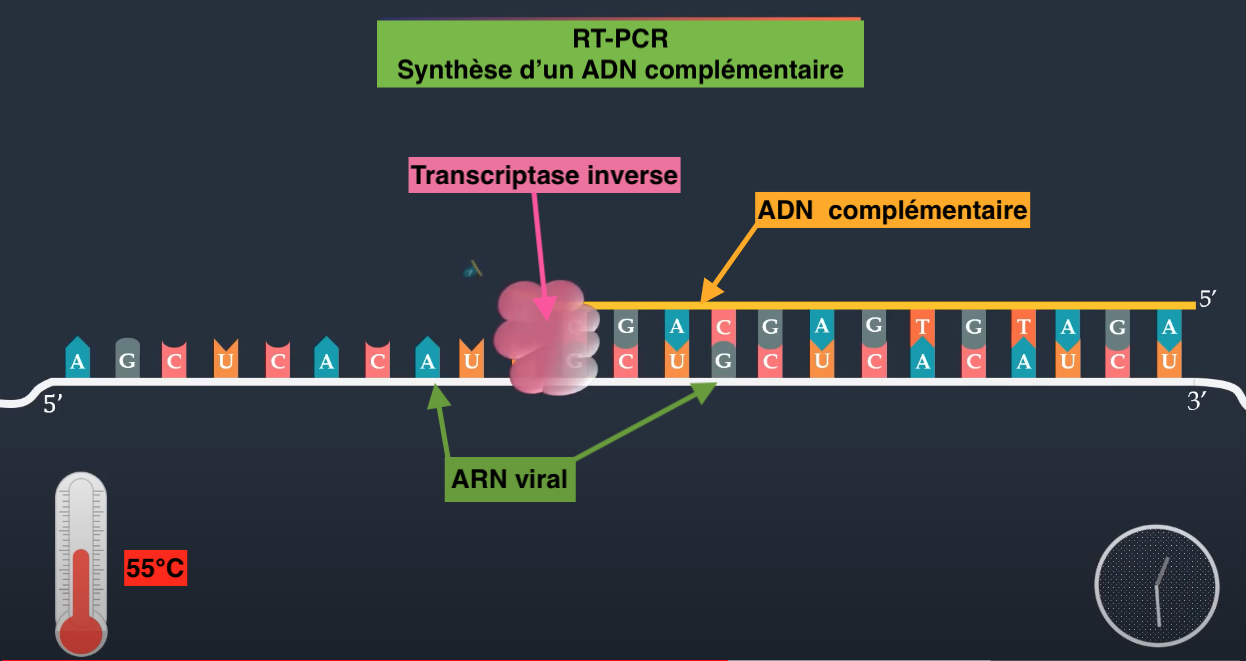
Après synthèse complète de l’ADN complémentaire, la température est portée à 95°C afin de le dissocier de l’ARN.
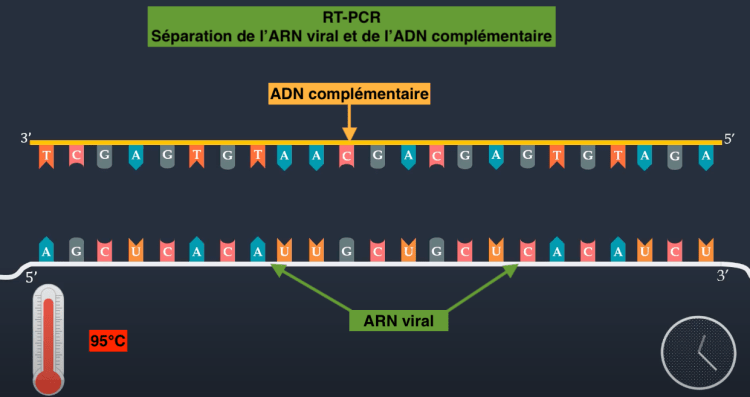
L’ADN complémentaire obtenu va servir de matrice pour reconstituer une double chaîne d’ADN à partir de laquelle la réaction classique de PCR va pouvoir démarrer.
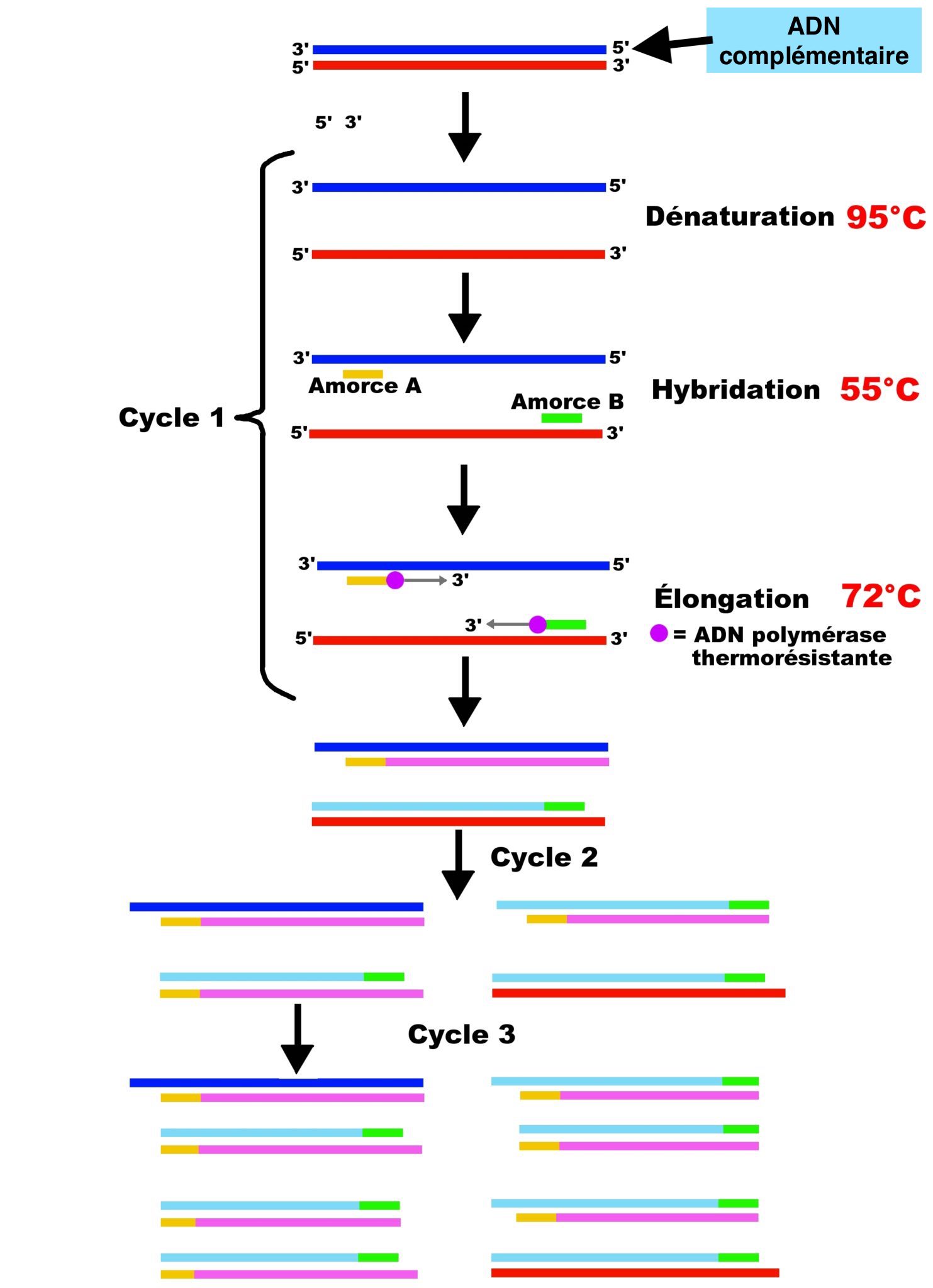
PCR à partir de l’ADN complémentaire de l’ARN viral
Depuis la publication initiale de Kary Mullis en 1985, de nombreuses adaptations de la PCR ont été réalisées. Parmi ces évolutions figurent notamment les PCR multiplexes, les PCR quantitatives et les PCR numériques.
PCR multiplexe
Lors d’une PCR multiplexe on utilise plusieurs couples d’amorces pour amplifier simultanément plusieurs segments d’ADN en une seule réaction.
Coronavirus et autres pathogènes
Les tests multiplexes sont utilisés pour la détection des mutations multiples des variants du SARS-CoV-2. Ces tests ciblent plusieurs gènes et visent les zones les moins susceptibles de muter.
D’autres tests de PCR multiplexe permettent d’effectuer le diagnostic de différentes maladies infectieuses.
Ainsi BioFire, filiale américaine de bioMérieux spécialisée en biologie moléculaire, a mis au point un test multiplexe permettant de détecter et d’identifier en 45 minutes 23 microbes différents (19 virus, 4 bactéries) responsables d’infections respiratoires aigües, Sars-cov-2 compris.
En effet, dans le cas des maladies respiratoires, les virus du Covid-19 et d’autres microbes donnent un tableau clinique semblable, marqué par des symptômes où l’on trouve de la fièvre, des courbatures, une fatigue généralisée, une toux, un essoufflement… ce qui rend le diagnostic difficile.

PCR multiplexe BioFire pour infections respiratoires
Cette même société a également mis au point un test PCR multiplexe pour identifier en moins d’une heure les 22 pathogènes (Bactéries, virus, parasites) les plus communément en cause dans les infections gastro-intestinales.

PCR multiplexe BioFire pour infections gastro-intestinales
Ce test est particulièrement intéressant pour détecter les maladies infectieuses d’origine alimentaire qui restent élevées en France. Une étude portant sur la période 2008-2013 fait état de 1,28 à 2,23 millions de cas annuels, dont 15 800 à 21 200 hospitalisations et entre 232 et 358 décès. Certains de ces pathogènes, comme les Escherichia coli produisant des Shigatoxines, provoquent le syndrome hémolytique et urémique (SHU) qui peut laisser de graves séquelles.
Ainsi, pour nombre de maladies infectieuses, les analyses par PCR multiplexe permettent de déterminer très rapidement la nature de l’agent microbien.
Police scientifique
Dans ce domaine on utilise systématiquement la PCR multiplexe car le nombre de séquences à amplifier est très important.
A titre d’exemple, l’ARN du coronavirus comporte 30 000 bases mais l’ADN humain en contient 3,2 milliards de paires. En conséquence, on amplifie actuellement entre 20 et 25 séquences en même temps avec des systèmes de PCR multiplexe dédiés à l’identification des personnes.
La figure ci-dessous constitue un exemple des zones (loci) analysées en identification humaine. Les numérotions de type D3, D16, D8 … correspondent aux chromosomes 3, 16, 8 … Les zones Y, A (amélogénine) permettent le sexage des individus.
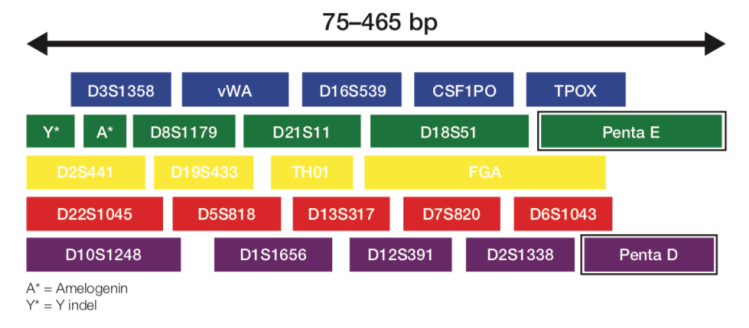
Loci analysés par PCR multiplexe pour l’identification humaine
Dans ce type d’analyse la majorité des fragments d’ADN amplifiés sont situés en dehors des gènes, c’est à dire dans les parties de l’ADN dites non codantes. Les gènes humains (ADN codant) ne représentent que 2% de l’ADN total.
PCR en temps réel ou quantitative
La PCR en temps réel, aussi appelée PCR quantitative, permet de déterminer la présence et la concentration d’une séquence d’ADN cible dans un échantillon. Cette technique quantitative utilise des sondes fluorescentes pour suivre, à chaque cycle, la quantité de produit formé. Elle peut être utilisée pour quantifier différentes cibles d’ADN en même temps, c’est à dire en multiplexe.
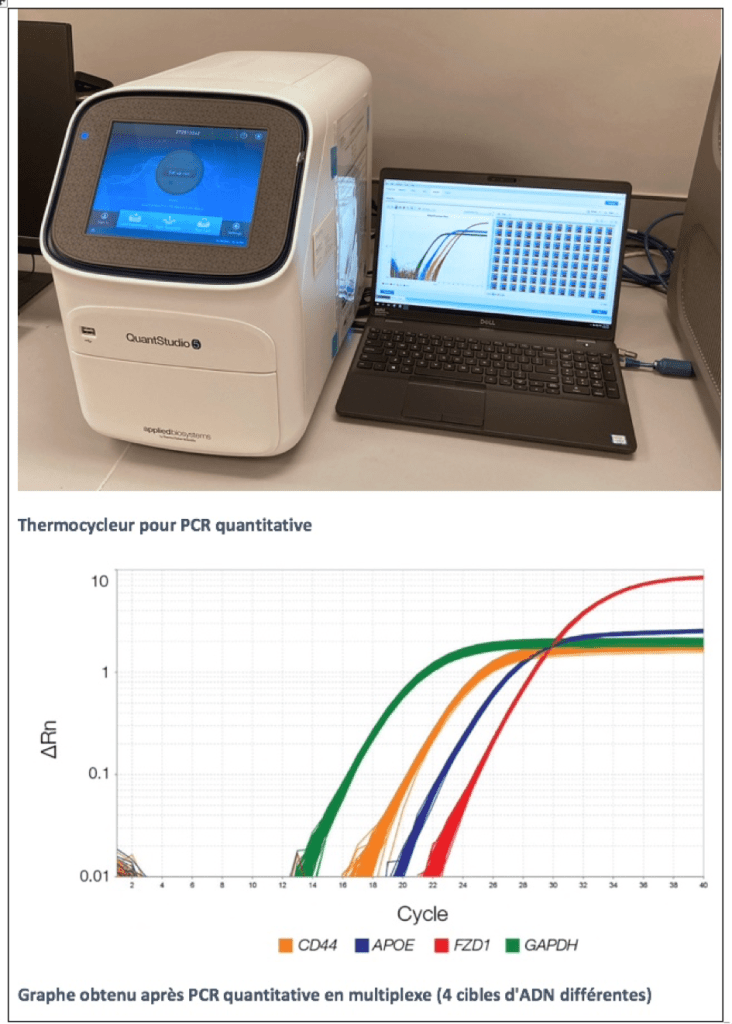
La RT- PCR multiplexe en temps réel est l’une des techniques les plus utilisées pour détecter les coronavirus.
Dans l’exemple présenté ci-dessous les flèches rouges indiquent les gènes du coronavirus qui vont être ciblés par les amorces. Généralement, le gène S qui porte l’information pour la pointe de fixation du virus (Protéine spike) n’est pas ciblé, car c’est le gène qui mute le plus.
Avec cette approche, les tests PCR peuvent convenir pour analyser plusieurs variants.
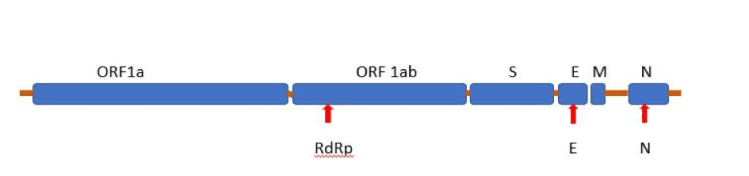
RdRp: gène de l’ARN polymérase virale
E: gène de la protéine d’enveloppe
N: gène de la nucléocapside
PCR numérique
Une nouvelle technique de PCR quantitative s’est développée ces dernières années grâce aux progrès de la microfluidique et de l’informatique, la PCR numérique. Le développement de cette technologie est le résultat des progrès spectaculaires réalisés en microfluidique (Nature 2006, 2012) et en informatique. La microfluidique permet d’accélérer la vitesse des réactions et de réduire la quantité de réactifs et d’échantillons.
PCR numérique en micro-chambres
La PCR en micro-chambres permet de réaliser des amplifications de l’ADN dans des volumes de l’ordre de quelques nanolitres (10-9 litre, soit un milliardième de litre).

Zooms successifs sur une plaque de 20 480 micro-chambres (Thermofisher)
Chaque micro-chambre est considérée comme un réacteur indépendant où peut se produire une amplification par PCR.
PCR numérique en microgouttelettes
Un deuxième type de PCR numérique utilise des microgouttelettes constituées d’une émulsion huile-eau. Ces gouttelettes permettent d’encapsuler les molécules d’ADN à amplifier et tous les réactifs nécessaires. Elle remplissent les mêmes fonctions que les plaques à 384 puits ou les micro-compartiments, mais dans un format encore plus petit permettant d’amplifier simultanément des milliers d’échantillons. Chaque gouttelette peut être considérée comme un micro-tube à essai dans lequel va se dérouler l’amplification par PCR. L’émission de fluorescence par les sondes associées aux amorces est ensuite mesurée.
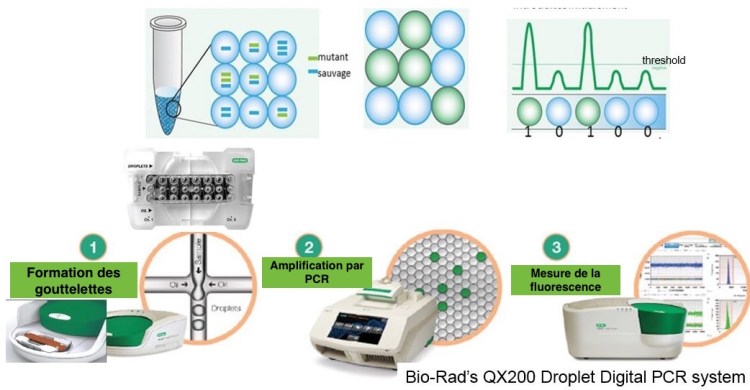
Appareillage de PCR numérique à microgouttelettes
Cette technique en microgouttelettes est notamment utilisée en cancérologie, dans la recherche d’ADN circulant d’origine tumorale, ainsi qu’en police scientifique.
Les prélèvements
Prélèvement nasopharyngé des cellules épithéliales
Pour la majorité des personnes, le prélèvement nasopharyngé à l’aide d’un écouvillon stérile constitue la première phase avant l’analyse par RT-PCR. Ce type de prélèvement permet de récupérer des cellules épithéliales qui tapissent les fosses nasales et constituent la porte d’entrée principale du virus dans les voies respiratoires
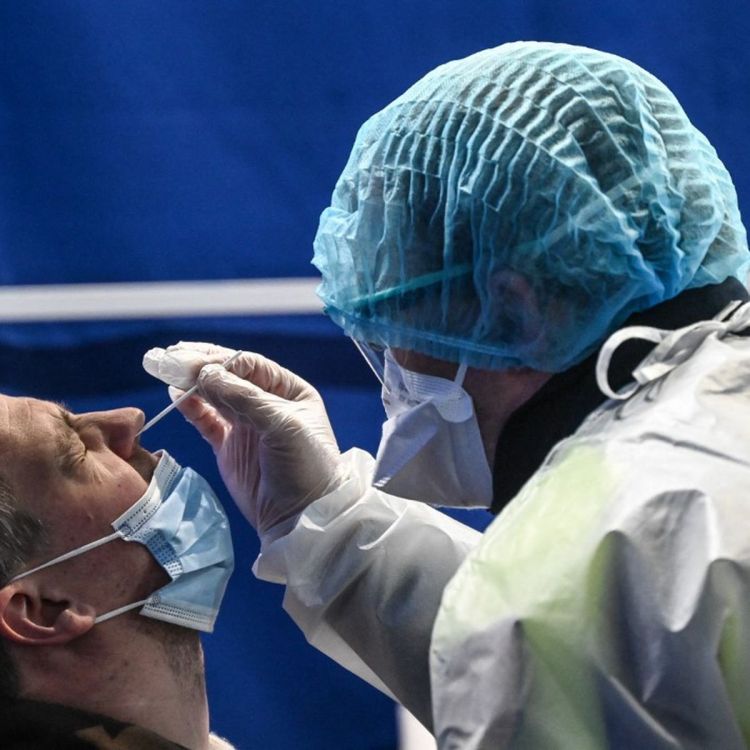
Prélèvement nasopharyngé
Les écouvillons sont acheminés en laboratoire et les cellules prélevées sont traitées pour libérer leur contenu, on parle alors d’extraction. Si de l’ARN viral est présent, il sera transcrit en un ADN spécifique qui sera amplifié lors de l’analyse RT-PCR précédemment décrite et le test sera dit positif.
Police scientifique
Les prélèvements biologiques réalisés en police scientifique se répartissent en deux grandes entités :
– Les prélèvements réalisés sur scène de crime, sur les lieux de catastrophes naturelles ou accidentelles comme les crashs aériens
– les prélèvements de cellules buccales sur individu
Sur scène de crime les traces biologiques recueillies peuvent être d’origines très diverses.

Prélèvements sur scène de crime
En France, les prélèvements buccaux issus des individus sont placés sur un support appelé FTA (Fast Technology Analysis). Il s’agit de cartes à base de cellulose enduites de détergents qui font éclater les cellules buccales et les microbes. Elles permettent la préservation et la fixation des ADN et des ARN ainsi libérés. On obtient alors un échantillon adapté à l’analyse de l’ADN par PCR et des ARN par RT-PCR sans risque de contamination pour les manipulateurs.
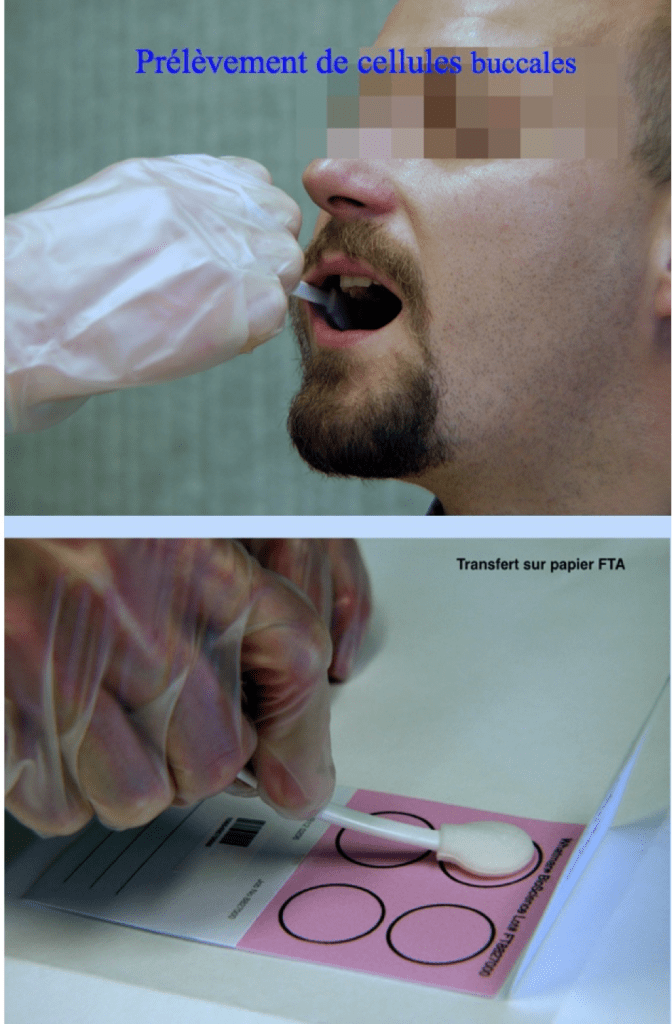
Ces supports facilitent la collecte, le transport, la purification et le stockage de l’ADN et de l’ARN sur le long terme et à température ambiante. Ils offrent également une compatibilité avec les systèmes robotisés dans le cadre des analyses PCR à haut débit. Au début des années 2000, en accord avec les représentants de la police et de la gendarmerie et dans la perspective de la mise en route du Fichier National des Empreintes Génétiques (FNAEG), j’avais donc préconisé leur utilisation en France pour tous les prélèvements sur individu.

In Police scientifique : Les experts au coeur de la scène de crime de Sébastien Aguilar, Benoit de Maillard, éditions Hachette 2017, p: 43
Automatisation des analyses
Depuis plusieurs années l’évolution des technologies fait partie intégrante de la réalité des laboratoires d’analyses.
Pour les laboratoires de biologie médicale, la pandémie due au COVID-19 a nécessité le traitement par PCR de très grandes quantités d’échantillons. De ce fait, le mouvement de regroupement des laboratoires de biologie médicale autour de plateaux techniques automatisés, qui était déjà amorcé, n’a fait que s’amplifier. Il en résulte que certains laboratoires de proximité ne sont plus que des sites de prélèvements.
En police scientifique, une unité automatisée d’analyse à haut débit des prélèvements sur individus à partir de papier FTA fonctionne au laboratoire de police scientifique de Lyon depuis 2006. Cette unité alimente le Fichier National d’Empreintes Génétiques (FNAEG).

Laboratoire de police scientifique de Lyon, unité robotisée pré-PCR